Referencje >> Microarray
Technologia Microarray
|
jak działa technologia Microarray?
|
identyfikacja bakterii za pomocą mikromacierzy
|
wykrywanie splicingu genów za pomocą mikromacierzy
|
Wprowadzenie do mikromacierzy
badania biologii molekularnej ewoluują poprzez rozwój technologii stosowanych do ich przenoszenia wynocha., Nie jest możliwe badanie dużej liczby genów przy użyciu tradycyjnych metod. DNA Microarray jest jeden taki technologia który umożliwia naukowcom badać i rozwiązywać problemy które kiedyś uważali być nie traceable. Można analizować ekspresję wielu genów w jednej reakcji szybko i efektywnie. Technologia DNA Microarray upoważniła społeczność naukową do zrozumienia podstawowych aspektów podkreślających wzrost i rozwój życia, a także do zbadania genetycznych przyczyn anomalii występujących w funkcjonowaniu ludzkiego ciała.,
typowy eksperyment microarray polega na hybrydyzacji cząsteczki mRNA do szablonu DNA, z którego pochodzi. Wiele próbek DNA jest używanych do budowy tablicy. Ilość mRNA związana z każdym miejscem na tablicy wskazuje poziom ekspresji różnych genów. Liczba ta może być liczona w tysiącach. Wszystkie dane są zbierane i generowany jest profil ekspresji genów w komórce.
Technika Microarray
tablica jest uporządkowanym układem próbek, w którym dopasowanie znanych i nieznanych próbek DNA odbywa się na podstawie zasad parowania., Eksperyment TABLICOWY wykorzystuje wspólne systemy oznaczania, takie jak mikropłytki lub standardowe membrany blotting. Rozmiary plamek próbki są zwykle mniejsze niż 200 mikronów średnicy zwykle zawierają tysiące plamek.
tysiące spotęgowanych próbek znanych jako sondy (o znanej tożsamości) unieruchamiane są na stałym podłożu (szkiełko mikroskopowe lub chipy silikonowe lub membrana nylonowa). Plamy mogą być DNA, cDNA lub oligonukleotydy. Są one używane do określenia komplementarnego wiązania nieznanych sekwencji, umożliwiając tym samym równoległą analizę ekspresji genów i odkrycia genów., Eksperyment z pojedynczym chipem DNA może dostarczyć informacji na temat tysięcy genów jednocześnie. Uporządkowane rozmieszczenie sond na podparciu jest ważne, ponieważ lokalizacja każdego miejsca na tablicy jest używana do identyfikacji genu.
rodzaje Mikroarray
w zależności od rodzaju unieruchomionej próbki używanej do konstruowania tablic i pobieranych informacji, eksperymenty Mikroarray można podzielić na trzy sposoby:
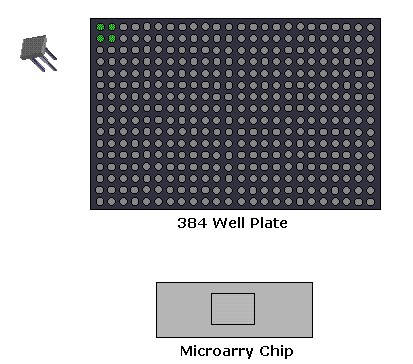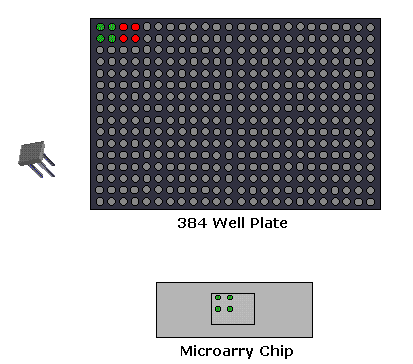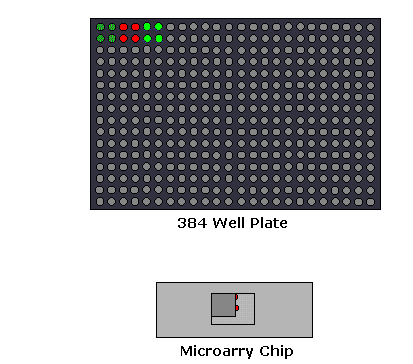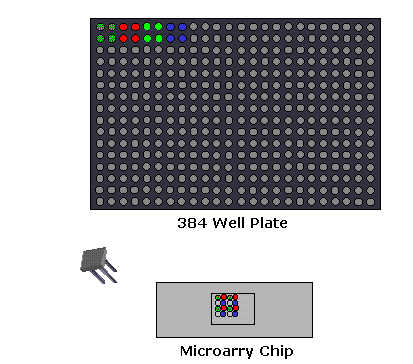 1., Analiza ekspresji Microarray: w ten eksperymentalny setup, cDNA czerpiący z mRNA znanych genów jest unieruchomiony. Próbka ma geny zarówno z normalnych, jak i chorych tkanek. Plamy o większej intensywności uzyskuje się dla chorego genu tkankowego, jeśli gen jest powyżej wyrażony w chorym stanie. Ten wzór ekspresji jest następnie porównywany do wzoru ekspresji genu odpowiedzialnego za chorobę.
1., Analiza ekspresji Microarray: w ten eksperymentalny setup, cDNA czerpiący z mRNA znanych genów jest unieruchomiony. Próbka ma geny zarówno z normalnych, jak i chorych tkanek. Plamy o większej intensywności uzyskuje się dla chorego genu tkankowego, jeśli gen jest powyżej wyrażony w chorym stanie. Ten wzór ekspresji jest następnie porównywany do wzoru ekspresji genu odpowiedzialnego za chorobę.
2. Microarray do analizy mutacji: do tej analizy naukowcy używają gDNA. Geny mogą różnić się od siebie tak mniej jak pojedyncza baza nukleotydów.,
pojedyncza różnica zasad między dwoma sekwencjami jest znana jako polimorfizm pojedynczego nukleotydu (SNP), a ich wykrywanie jest znane jako wykrywanie SNP.
3. Porównawcza hybrydyzacja Genomowa: służy do identyfikacji w zwiększaniu lub zmniejszaniu ważnych fragmentów chromosomów zawierających geny zaangażowane w chorobę.
zastosowania Microarrays
Gene Discovery: DNA Microarray technologia pomaga w identyfikacji nowych genów, wiedzieć o ich funkcjonowaniu i poziomie ekspresji w różnych warunkach.,
diagnoza choroby: technologia DNA Microarray pomaga naukowcom dowiedzieć się więcej o różnych chorobach, takich jak choroby serca, choroby psychiczne, choroby zakaźne, a zwłaszcza badania nad rakiem. Do niedawna, różne rodzaje raka zostały sklasyfikowane na podstawie narządów, w których nowotwory rozwijają. Teraz, wraz z rozwojem technologii microarray, naukowcy będą mogli dalej klasyfikować rodzaje raka na podstawie wzorców aktywności genów w komórkach nowotworowych., To ogromnie pomoże społeczności farmaceutycznej do opracowania bardziej skutecznych leków, jak strategie leczenia będą ukierunkowane bezpośrednio do określonego rodzaju raka.
odkrycie leku: Technologia Microarray ma szerokie zastosowanie w Farmakogenomice. Farmakogenomika jest badaniem korelacji między odpowiedziami terapeutycznymi na leki a profilami genetycznymi pacjentów. Analiza porównawcza genów z chorej i normalnej komórki pomoże zidentyfikować biochemiczną konstytucję białek syntetyzowanych przez chore geny., Naukowcy mogą wykorzystać te informacje do syntezy leków, które zwalczają te białka i zmniejszają ich działanie.
badania toksykologiczne: Technologia Microarray zapewnia solidną platformę do badań wpływu toksyn na komórki i ich przekazywania potomstwu. Toksykogenomika ustanawia korelację między reakcjami na toksyny a zmianami w profilach genetycznych komórek narażonych na takie toksyny.
GEO
w niedawnej przeszłości technologia microarray była szeroko stosowana przez społeczność naukową., W związku z tym, na przestrzeni lat, nie było wiele generacji danych związanych z ekspresją genów. Dane te są rozproszone i nie są łatwo dostępne do użytku publicznego. W celu ułatwienia dostępu do tych danych, National Center for Biotechnology Information (NCBI) sformułował ekspresję genów Omnibus lub GEO. Jest to obiekt repozytorium danych, który zawiera dane na temat ekspresji genów z różnych źródeł.,v id=”0052483fe5″>
Other Parameters
-
Probe Location
1., 3 'end bias: oligos wybrany powinien być w kierunku 3′ end genu, tj. domyślnie: 3 ' end.
2 . Oligos powinien być zaprojektowany domyślnie w granicach 999 podstaw 3 ' końca. Zakres może wynosić od 0 do 1500 baz.
-
oligosy powinny być wolne od homologii krzyżowej (tzn. powinny być przeszukiwane według odpowiedniej kategorii genomu).