Wie microarray technologie funktioniert?
|
Bakterielle Identifizierung mittels Mikroarrays
|
Genspleißerkennung mittels Mikroarrays
|
Einführung in Microarray
Die molekularbiologische Forschung entwickelt sich durch die Entwicklung der Technologien, mit denen sie durchgeführt werden., Es ist nicht möglich, eine große Anzahl von Genen mit traditionellen Methoden zu erforschen. DNA Microarray ist eine solche Technologie, die es den Forschern ermöglicht, Probleme zu untersuchen und anzugehen, die einst als nicht rückverfolgbar galten. Man kann die Expression vieler Gene in einer einzigen Reaktion schnell und effizient analysieren. Die DNA-Mikroarraytechnologie hat die wissenschaftliche Gemeinschaft befähigt, die grundlegenden Aspekte des Wachstums und der Entwicklung des Lebens zu verstehen und die genetischen Ursachen von Anomalien im menschlichen Körper zu untersuchen.,
Ein typisches Mikroarray-Experiment beinhaltet die Hybridisierung eines mRNA-Moleküls mit der DNA-Vorlage, aus der es stammt. Viele DNA-Proben werden verwendet, um ein Array zu konstruieren. Die Menge an mRNA, die an jede Stelle im Array gebunden ist, gibt das Expressionsniveau der verschiedenen Gene an. Diese Zahl kann in Tausenden laufen. Alle Daten werden gesammelt und ein Profil für die Genexpression in der Zelle generiert.
Mikroarray-Technik
Ein Array ist eine geordnete Anordnung von Proben, bei der die Übereinstimmung bekannter und unbekannter DNA-Proben basierend auf Basenpaarungsregeln erfolgt., Ein Array-Experiment nutzt gängige Assay-Systeme wie Mikroplatten oder Standard-Blotting-Membranen. Die Probenfleckgrößen sind typischerweise weniger als 200 Mikrometer im Durchmesser enthalten in der Regel Tausende von Flecken.
Tausende von gefleckten Proben, die als Sonden (mit bekannter Identität) bekannt sind, werden auf einem festen Träger (Mikroskopglasschieber oder Siliziumchips oder Nylonmembran) immobilisiert. Die Flecken können DNA, cDNA oder Oligonukleotide sein. Diese werden verwendet, um die komplementäre Bindung der unbekannten Sequenzen zu bestimmen und so eine parallele Analyse für die Genexpression und Genentdeckung zu ermöglichen., Ein Experiment mit einem einzelnen DNA-Chip kann gleichzeitig Informationen über Tausende von Genen liefern. Eine geordnete Anordnung der Sonden auf dem Träger ist wichtig, da die Position jedes Punktes auf dem Array zur Identifizierung eines Gens verwendet wird.
Typen von Mikroarrays
Abhängig von der Art der immobilisierten Probe, die für Arrays verwendet wird, und den abgerufenen Informationen können die Mikroarrayexperimente auf drei Arten kategorisiert werden:
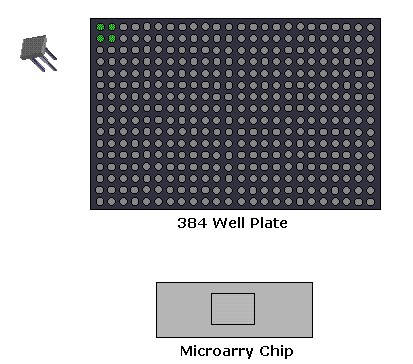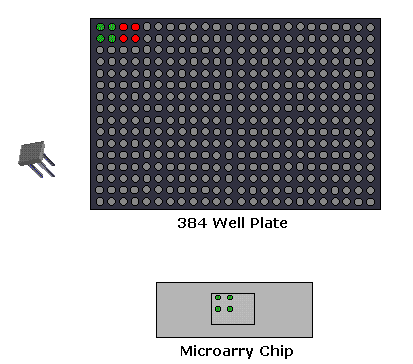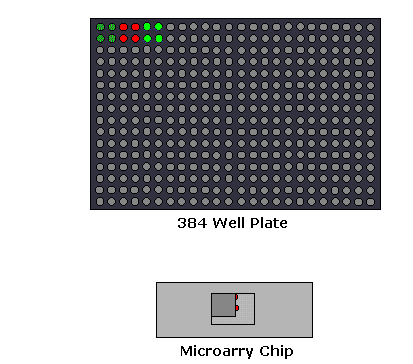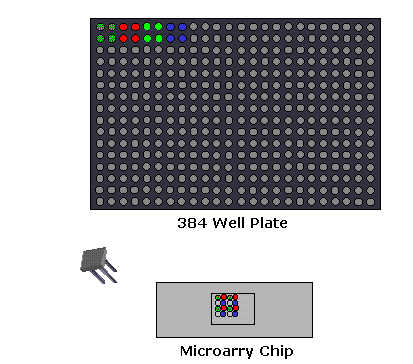 1., Mikroarray-Expressionsanalyse: In diesem Versuchsaufbau wird die aus der mRNA bekannter Gene abgeleitete cDNA immobilisiert. Die Probe hat Gene sowohl aus dem normalen als auch aus dem erkrankten Gewebe. Flecken mit größerer Intensität werden für das erkrankte Gewebegen erhalten, wenn das Gen im erkrankten Zustand überexprimiert wird. Dieses Expressionsmuster wird dann mit dem Expressionsmuster eines für eine Krankheit verantwortlichen Gens verglichen.
1., Mikroarray-Expressionsanalyse: In diesem Versuchsaufbau wird die aus der mRNA bekannter Gene abgeleitete cDNA immobilisiert. Die Probe hat Gene sowohl aus dem normalen als auch aus dem erkrankten Gewebe. Flecken mit größerer Intensität werden für das erkrankte Gewebegen erhalten, wenn das Gen im erkrankten Zustand überexprimiert wird. Dieses Expressionsmuster wird dann mit dem Expressionsmuster eines für eine Krankheit verantwortlichen Gens verglichen.
2. Microarray zur Mutationsanalyse: Für diese Analyse verwenden die Forscher gDNA. Die Gene können sich um so weniger voneinander unterscheiden wie eine einzelne Nukleotidbasis.,
Eine einzelne Basendifferenz zwischen zwei Sequenzen wird als Einzelnukleotidpolymorphismus (SNP) bezeichnet und deren Detektion wird als SNP-Detektion bezeichnet.
3. Vergleichende genomische Hybridisierung: Sie dient zur Identifizierung der Zunahme oder Abnahme der wichtigen Chromosomenfragmente, die Gene enthalten, die an einer Krankheit beteiligt sind.
Anwendungen von Mikroarrays
Gen Entdeckung: DNA Microarray technologie hilft in die identifizierung von neue gene, wissen über ihre funktion und expression ebenen unter verschiedenen bedingungen.,
Krankheitsdiagnose: Die DNA-Mikroarraytechnologie hilft Forschern, mehr über verschiedene Krankheiten wie Herzkrankheiten, psychische Erkrankungen, Infektionskrankheiten und insbesondere das Studium von Krebs zu erfahren. Bis vor kurzem wurden verschiedene Krebsarten anhand der Organe klassifiziert, in denen sich die Tumoren entwickeln. Mit der Entwicklung der Microarray-Technologie wird es den Forschern nun möglich sein, die Krebsarten anhand der Muster der Genaktivität in den Tumorzellen weiter zu klassifizieren., Dies wird der pharmazeutischen Gemeinschaft enorm helfen, wirksamere Medikamente zu entwickeln, da die Behandlungsstrategien direkt auf die spezifische Krebsart abzielen.
Wirkstoffentdeckung: Die Microarray-Technologie findet umfangreiche Anwendung in der Pharmakogenomik. Pharmakogenomik ist die Untersuchung von Korrelationen zwischen therapeutischen Reaktionen auf Medikamente und den genetischen Profilen der Patienten. Die vergleichende Analyse der Gene einer erkrankten und einer normalen Zelle hilft bei der Identifizierung der biochemischen Konstitution der von den erkrankten Genen synthetisierten Proteine., Die Forscher können diese Informationen verwenden, um Medikamente zu synthetisieren, die mit diesen Proteinen bekämpfen und ihre Wirkung reduzieren.
Toxikologische Forschung: Die Microarray-Technologie bietet eine robuste Plattform für die Erforschung der Auswirkungen von Toxinen auf die Zellen und deren Weitergabe an die Nachkommen. Die Toxikogenomik stellt eine Korrelation zwischen den Reaktionen auf toxische Substanzen und den Veränderungen der genetischen Profile der Zellen her, die solchen toxischen Substanzen ausgesetzt sind.
GEO
In der jüngeren Vergangenheit wurde die Microarray-Technologie von der wissenschaftlichen Gemeinschaft weitgehend genutzt., Folglich gab es im Laufe der Jahre viele Daten im Zusammenhang mit der Genexpression. Diese Daten sind verstreut und nicht leicht für die öffentliche Nutzung verfügbar. Um den Zugang zu diesen Daten zu erleichtern, hat das National Center for Biotechnology Information (NCBI) den Genexpressions-Omnibus oder GEO formuliert. Es ist eine Daten-Repository-Anlage, die Daten über die Genexpression aus verschiedenen Quellen enthält.,v id=“0052483fe5″>
| Parameter |
Minimum Value |
Maximum Value |
Default Value |
Unit |
| Probe Length |
10
|
99
|
30
|
bases
|
| Probe Length tolerance |
0
|
15
|
3
|
|
| Probe Target Tm |
40
|
99
|
63
|
°C
|
| Probe Tm Tolerance (+) |
0.,1
|
99
|
5
|
|
| Haarnadel Max ÄG |
0,1
|
99,9
|
4
|
cal/mol
|
| Self Dimer ÄG |
0.1
|
99.,v id=“0052483fe5″>
|
| Parameter |
Minimum Value |
Maximum Value |
Default Value |
Unit |
| Probe Length |
10
|
99
|
40
|
bases
|
| Probe Length tolerance |
0
|
15
|
3
|
|
| Probe Target Tm |
40
|
99
|
70
|
°C
|
| Probe Tm Tolerance (+) |
0.,1
|
99
|
5
|
|
| Haarnadel Max ÄG |
0,1
|
99,9
|
6
|
|
| Self Dimer ÄG |
0.1
|
99.,/div>
| Parameter |
Minimum Value |
Maximum Value |
Default Value |
Unit |
| Probe Length |
10
|
99
|
70
|
bases
|
| Probe Length tolerance |
0
|
15
|
3
|
|
| Probe Target Tm |
40
|
99
|
75
|
°C
|
|
Probe Tm Tolerance (+/- above)
|
0.,1
|
99
|
5
|
|
| Hairpin Max ÄG |
0.1
|
99.9
|
6
|
Kcal/mol
|
| Self Dimer ÄG |
0.1
|
99.9
|
8
|
Kcal/mol
|
| Run/Repeat |
2
|
99
|
6
|
bases
|
|
 1., Mikroarray-Expressionsanalyse: In diesem Versuchsaufbau wird die aus der mRNA bekannter Gene abgeleitete cDNA immobilisiert. Die Probe hat Gene sowohl aus dem normalen als auch aus dem erkrankten Gewebe. Flecken mit größerer Intensität werden für das erkrankte Gewebegen erhalten, wenn das Gen im erkrankten Zustand überexprimiert wird. Dieses Expressionsmuster wird dann mit dem Expressionsmuster eines für eine Krankheit verantwortlichen Gens verglichen.
1., Mikroarray-Expressionsanalyse: In diesem Versuchsaufbau wird die aus der mRNA bekannter Gene abgeleitete cDNA immobilisiert. Die Probe hat Gene sowohl aus dem normalen als auch aus dem erkrankten Gewebe. Flecken mit größerer Intensität werden für das erkrankte Gewebegen erhalten, wenn das Gen im erkrankten Zustand überexprimiert wird. Dieses Expressionsmuster wird dann mit dem Expressionsmuster eines für eine Krankheit verantwortlichen Gens verglichen.