Krebs bezieht sich auf eine komplexe und heterogene Gruppe von Krankheiten, die durch die unkontrollierte und ungeordnete Proliferation von Zellen gekennzeichnet sind, die häufig in andere Gewebe eindringen können. Krebs entsteht normalerweise in somatischen Zellen, die sich als Folge einer Reihe genetischer Mutationen den Mechanismen entziehen, die die Gewebehomöostase regulieren, wie Zell-zu-Zell-Kontakthemmung, Differenzierungssignale und Zelltodinduktion., Die Mutationen, die für die Tumortransformation verantwortlich sind, betreffen zwei Hauptgruppen von Genen, die als Proto-Onkogene (Stimulatoren des Zellzyklus) und Tumorsuppressoren (Repressoren der Zellzyklusprogression) bekannt sind. Diese funktionellen Veränderungen können als Folge von einzelnen Nukleotidmutationen auftreten, können aber auch durch größere Modifikationen des genetischen Materials verursacht werden, wie Insertionen, Deletionen, Duplikationen oder Translokationen eines Chromosomenfragments. Diese Anomalien in Krebszellen können als Tumorbiomarker verwendet werden., Die Quantifizierung von Veränderungen in der Anzahl der Genkopien oder Gen-Re-Arrangements ist entscheidend für unser Verständnis der Tumorbiologie, daher die Bedeutung von Gentests auf der Grundlage molekularer Zytogenetik Profiling.
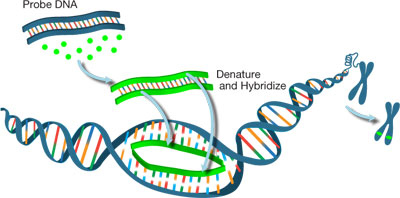
Abbildung 1: Kennzeichnung DNA-Sonden für FISH-Analyse.
Was ist FISCH ?
Fluoreszenz-in-situ-Hybridisierung (FISH) ist eine zytogenetische Technik, die zum Nachweis und zur Lokalisierung von Nukleotidsequenzen (DNA oder RNA) in Geweben oder Zellen verwendet wird., FISCHE können zum „Mapping“ des genetischen Materials in menschlichen Zellen verwendet werden, um Informationen über den Ort, die Länge und die Anzahl der Kopien bestimmter Gene oder Chromosomenanteile bereitzustellen. Es erfordert die Synthese einer fluoreszenzmarkierten Sonde, bei der es sich um eine Nukleinsäuresequenz handelt, die an ein Fluoreszenzreportermolekül gebunden ist und dann an eine bestimmte Zielsequenz bindet (hybridisiert). Fischsonden können mit verschiedenen Methoden (z. B. Nick-Translation, zufällige Grundierung) markiert werden, ausgehend von verschiedenen Nukleinsäureingaben, wie genomischer DNA/RNA, bakteriellen künstlichen Chromosomen (BACs) oder Cosmiden.,
Der erste Schritt im FISH-Prozess ist die Immobilisierung von Metaphase-chromosomalen Spreads oder Interphasenzellen, die die Ziel-DNA auf einem Glasschieber enthalten. Als nächstes werden sowohl die DNA-Probe als auch die Fischsonde durch Hitze denaturiert. Wenn die Sonde mit dem genetischen Zielmaterial in Kontakt kommt, bindet sie spezifisch an ihre komplementäre Sequenz auf dem Chromosom. Hybride, die zwischen den Sonden und ihren Sequenzzielen gebildet werden, können dann mit einem Fluoreszenzmikroskop visualisiert werden (Abbildung 1)., Im Allgemeinen können zwei Arten von Fischsonden unterschieden werden: die Chromosomenaufzählungssonden (CEPs oder ZENs), die auf die perizentromeren Regionen des Chromosoms abzielen und zur Aufzählung von Chromosomen verwendet werden; und lokusspezifische Indikatoren (LSI), die spezifisch Gene von Interesse erkennen.
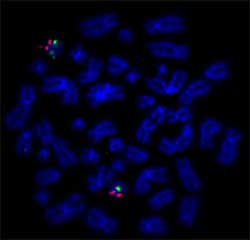
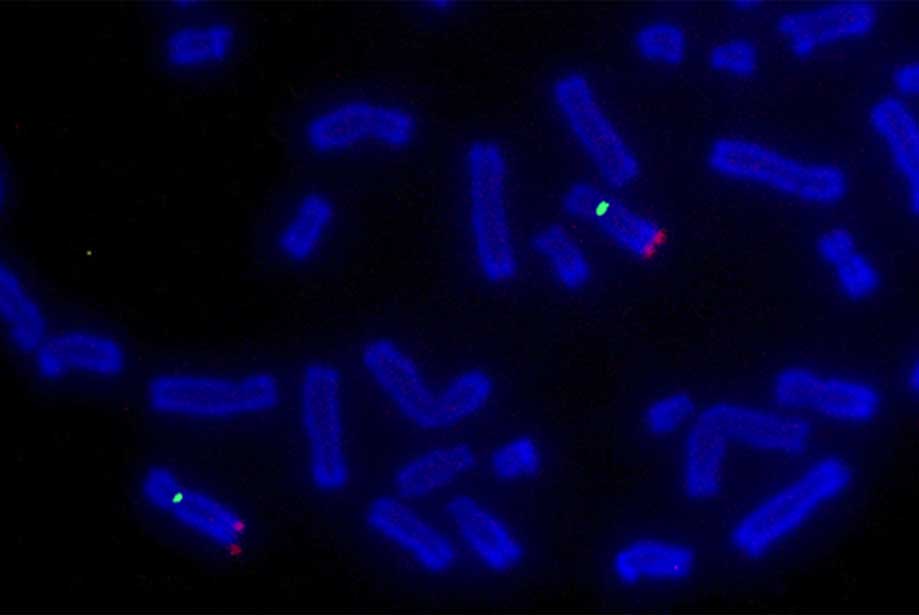
Abbildung 2: Beispiel FISCH Ergebnisse: Nick Translation DNA Labeling System 2.0 wurde verwendet, um label BAC DNA-Sonde für TP53 mit SEEBRIGHT® Orange 552 dUTP-und BAC-DNA-Sonde für das Centromer 17 mit SEEBRIGHT® Green 496 dUTP., Markierte Sonden wurden zu Metaphase-Spreads hybridisiert. (Institut Universitaire du Cancer Toulouse Oncopole)
FISCH hat mehrere Vorteile gegenüber anderen klassischen zytogenetischen Techniken, wie die G-banding-karyotypisierung her. Erstens hat es eine höhere Auflösung (20-150kb vs 5Mb). Darüber hinaus kann FISCH sowohl auf Metaphase-als auch auf Interphasenchromosomen angewendet werden, was bedeutet, dass Zellen mehrere Tage lang nicht kultiviert werden müssen, bevor Chromosomen für die Analyse vorbereitet werden können., Dies impliziert auch, dass FISCH für die Analyse verschiedener Arten von Probentypen geeignet ist, einschließlich fester Tumoren und formalinfixierter Paraffin eingebetteter (FFPE) Gewebe. Darüber hinaus können Fischsonden mit verschiedenen Fluorophoren beschriftet werden, was die gleichzeitige Überwachung mehrerer Standorte ermöglicht.
FISH-ing Chromosomenaberrationen bei Krebs
FISCH kann dank seiner Vielseitigkeit sowohl für die zytogenetische Analyse fester Tumoren (z. B. Brustkrebs, nichtkleinzelliger Lungenkrebs, Darmkrebs) als auch für hämatologische oder Blutkrebserkrankungen (z. B. Leukämie, Lymphome, multiples Myelom) verwendet werden., Der Nachweis genetischer Anomalien ist nicht nur für Krebs nützlich, sondern auch als Instrument zur Analyse genetischer Prädisposition und krankheitsspezifischer Informationen sowie zur Vorhersage eines chemotherapeutischen Ergebnisses.
FISCH für Lungenkrebs
Lungenkrebs ist die am häufigsten diagnostizierte und die häufigste Ursache für krebsbedingte Todesfälle. Insbesondere der nicht-kleinzellige Lungenkrebs (NSCLC) macht ~80-85% aller Lungenkrebserkrankungen aus. Somatische Mutationen an EGFR-und ALK-Genen sind häufig mit NSCLC assoziiert., EGFR (Epidermal Growth Factor Receptor) ist eine Klasse von Tyrosinkinase-Rezeptoren, deren Aktivität bei verschiedenen Arten von epithelialen Malignomen (einschließlich Lungenkrebs) dereguliert ist, da sie eine wichtige Rolle bei der Proliferation, Angiogenese und Metastasierung von Krebszellen spielen. Aus diesem Grund werden häufig unterschiedliche Strategien zur Störung der EGFR-Funktion für die Therapie von Patienten ausgenutzt. Inhibitoren der Tyrosinkinaseaktivität dieser Rezeptoren (z. B. Erlotinib, Gefitinib) werden häufig in klinischen NSCLC-Behandlungen eingesetzt., Leider sind einige Patienten aufgrund der Vielzahl genetischer Mutationen, die der EGFR-Dysfunktion zugrunde liegen, unempfindlich gegen diese Art der Behandlung. Verschiedene Patientengruppen können in der Tat anhand der Art der Veränderung durch EGFR unterschieden werden, z. B. Genamplifikation, Deletion oder einzelne Nukleotidsubstitutionen, die ihre Aktivität auf unterschiedliche Weise verändern können (dh nicht über die Tyrosinkinasedomäne). Infolgedessen ist die von FISH ermittelte EGFR-Kopiernummer einer der Biomarker, die zur Auswahl der richtigen Therapie verwendet werden.,
FISCH wird häufig verwendet, um Inversionen oder Translokationen im ALK-Gen zu erkennen. Das ALK-Gen befindet sich auf dem kurzen Arm von Chromosom 2 (2p23) und kodiert für den Transmembran-Tyrosinkinase-Rezeptor. ALK sollte nicht in der erwachsenen Lunge ausgedrückt werden. Unter pathologischen Bedingungen bricht das ALK-Gen jedoch und verschmilzt sein 3′ (das die Tyrosinkinase-Domäne enthält) mit dem 5′ anderer Gene. Dieses Ereignis kann zur unkontrollierten Aktivierung von ALK-Downstream-Signalwegen führen. Die häufigste Fusion tritt bei EML4 aufgrund einer Inversion am kurzen Arm von Chromosom 2 auf.,
FISH ist die von der FDA zugelassene Methode zum Nachweis von Inversionen oder Translokationen im ALK-Gen. Typischerweise werden Sonden, die auf die 3′ – und die 5′ – Region des Gens abzielen, mit verschiedenen Fluorophoren markiert: In negativen Kernen erscheinen die Farben nahe beieinander (oft überlappend), während sich die Signale in Krebszellen infolge der Chromosomenumlagerung aufspalten (Abbildung 4).

Abbildung 3: Repräsentative Ansicht von ALK-gen-Translokation nachgewiesen durch FISH., 5′ und 3′ Regionen des Gens werden mit einer grünen bzw. einer roten Fluoreszenz visualisiert. A. Wilde Art Kern. B. Krebszellkern, zeigt die charakteristischen Sonden gespalten. C. Krebs zellkern, zeigt die charakteristischen sonden split. Eine dritte Sonde kann verwendet werden, um die tatsächlich auftretende Chromosomenumlagerung zu definieren.
Eine dritte Sonde, die auf den potenziellen ALK-Partner abzielt, könnte die bei einem Patienten auftretende Chromosomenumlagerung weiter bestätigen und definieren (Abbildung 4C)., Lungenkrebs mit Mutationen, die zu einer ALK-Hyperaktivierung führen, kann mit ALK-Inhibitoren wie Crizotinib behandelt werden.
FISCH für Brustkrebs
Dies ist die häufigste Malignität bei Frauen und die zweithäufigste Ursache für krebsbedingten Tod weltweit. Brustkrebs ist häufig durch Abnormalitäten im Rezeptorstatus gekennzeichnet, die zu einer Hochregulierung zellulärer Transduktionswege führen, die für die Zellproliferation und das Überleben verantwortlich sind. Insbesondere ist bekannt, dass etwa 20-30% der Brustkrebstumoren HER2/Neu, ein Mitglied der EGFR-Familie, überexprimieren., Eine häufige Behandlung in diesen Fällen ist Trastuzumab, ein humanisierter monoklonaler Antikörper, der 1998 von der FDA zur Behandlung von Brustkrebs zugelassen wurde. Sein genauer molekularer Mechanismus muss noch geklärt werden, aber dieser Antikörper verhindert wahrscheinlich die HER2-Aktivierung durch Bindung an seine extrazelluläre Domäne. Darüber hinaus scheint es eine Tumorzelllyse zu induzieren, die die antikörperabhängige Zellzytotoxizität (ADCC) stimuliert. FISCHE, die eine geeignete Sonde gegen HER2 verwenden, können verwendet werden, um zusätzliche Kopien des Gens zu identifizieren, ein Zeichen dafür, dass es eher auf die Behandlung mit Trastuzumab anspricht.,
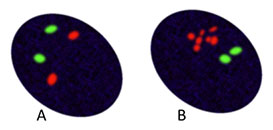
Abbildung 4: Repräsentative Ansicht von möglichen Brust-Karzinom-Zellen. HER2-Signal wird in rot dargestellt; Centromer 17 Sonde (grün) kann verwendet werden, um Chromosomennummer aufzuzählen. A. HER2-nicht amplifiziertes Brustkarzinom: zwei Zentromer 17 und zwei Kopien des HER2-Gens wie erwartet. B. HER2-amplifizierter Brustkarzinom-Mehrfachnachweis für HER2.
FISCH für Blasenkrebs
Es ist die fünfthäufigste menschliche Malignität und der zweithäufigste diagnostizierte Urogenitaltumor nach Prostatakrebs., Es ist eine polygene Erkrankung, was bedeutet, dass sie mit mehreren genetischen Anomalien in Verbindung gebracht wurde, wie Mutationen in FGFR3 -, RB1 -, HRAS -, TP53-oder TSC1-Genen. Das Initiierungsereignis ist jedoch wahrscheinlich eine Mutation in der 9p21-Region, die das p16/CDKN2A-Gen enthält. Darüber hinaus sind Blasenkrebszellen durch einen erhöhten Grad an Chromosomeninstabilität (CIN) gekennzeichnet. Dysfunktionelle Chromosomenverdopplung oder-segregation während der Mitose verursachen DNA-Umlagerungen und-translokationen, den Gewinn oder Verlust ganzer Chromosomen (Aneuploidie) oder Chromosomenfragmente., Die resultierende Unwucht des genetischen Materials verschlechtert sich nach jedem Zellzyklus. Die daraus resultierenden genomischen Muster können mit den verschiedenen Stadien der Tumorentwicklung zusammenhängen, wobei die invasiveren Formen die höhere Anzahl zytogenetischer Veränderungen aufweisen.
Die numerischen und strukturellen chromosomalen Veränderungen in Blasenkrebszellen können als Tumormarker verwendet werden.
Konkret ist der gleichzeitige Nachweis der Variation der Kopienzahl der Chromosomen 3, 7 und 17 und die Deletion der 9p21-Region (mit p16) durch FISCHE mit vier verschiedenen Sonden eine gängige Praxis., Diese Methode ist nützlich, um Informationen über das Fortschreiten und Wiederauftreten von Krebs bereitzustellen.
FISCH für chronische lymphatische Leukämie (CLL)
Die Analyse hämatologischer Malignome ist eines der typischen Beispiele für die Vorteile von FISCHEN für die Analyse von Proben, die durch einen variablen Karyotyp und eine geringe mitotische Aktivität gekennzeichnet sind. Die CLL ist die häufigste Leukämie bei Erwachsenen. Es wurde nicht mit einer spezifischen wiederkehrenden genetischen Veränderung in Verbindung gebracht., Stattdessen wurde, ähnlich wie bei Blasenkrebs, Ein Panel verschiedener Mutationen mit verschiedenen Schweregraden der Erkrankung in Verbindung gebracht und als prädiktive Indikatoren für den klinischen Verlauf des Patienten verwendet. Fischpaneele enthalten in diesem Fall häufig Sonden zum Nachweis von Trisomie 12 und Deletionen 11q, 13q und 17p., Deletion 11q, die in den meisten Fällen das Gen ATM betrifft, wird bei Patienten gefunden, die ein schnelles Fortschreiten des Krebses zeigen; Trisomie 12 ist mit fortgeschrittenen Stadien der Krankheit, Resistenz gegen Chemotherapie und kürzeren Überlebenszeiten verbunden; Deletion 13q ist die am häufigsten gefundene und ist im Allgemeinen mit einer günstigeren Prognose verbunden; Deletion 17p beinhaltet oft eine Deletion des TP53-Gens und entspricht dem fortgeschrittenen Stadium des Tumors mit einer schlechten Überlebensrate.
Enzo Life Sciences ist ein weltweit führender Anbieter von DNA-und RNA-Labeling-Technologien., Wir bieten eine Reihe von Produkten für Ihre Bedürfnisse in der Genomik und Krebsforschung. Eine einfache und effiziente Methode zur Erzeugung markierter DNA finden Sie in unserem Nick translation DNA Labeling Kit sowie in einer Liste unserer SEEBRIGHT® fluorescent Dye-dUTPs und Allylamine-dUTP. Unsicher, ob Biotin oder Digoxigenin für die ISH-Sondenmarkierung verwendet werden soll? Wir können Ihnen dort helfen! Vergessen Sie nicht, Enzo ‚ s Spectra Viewer auf Anregung und Emissions-Wellenlängenprofile gängiger Farbstoffe und unserer anderen fluoreszenzzellbasierten Assays zu überprüfen., Während Sie dabei sind, lesen Sie bitte unsere TechNote zu den Vor-und Nachteilen von FISCH, aCGH und NGS sowie zur Verwendung von Allylamine-dUTP für die Kennzeichnung von FISCH-DNA-Sonden. Bei allen Fragen und Bedenken zu einem unserer Produkte steht Ihnen unser technisches Support-Team zur Verfügung.