referencias >> empalme de genes
resumen de empalme de genes & técnicas
Gen introducción
El Empalme de genes es una modificación post-transcripcional en la que un solo gen puede codificar múltiples proteínas. El empalme genético se realiza en eucariotas, antes de la traducción del ARNm, mediante la inclusión o exclusión diferencial de regiones de pre-ARNm. El empalme genético es una fuente importante de diversidad proteica., Durante un evento típico de empalme de genes, el pre-ARNm transcrito de un gen puede conducir a diferentes moléculas maduras de ARNm que generan múltiples proteínas funcionales. Por lo tanto, el empalme de genes permite que un solo gen aumente su capacidad de codificación, lo que permite la síntesis de isoformas de proteínas que son estructural y funcionalmente distintas. El empalme de genes se observa en una alta proporción de genes. En las células humanas, se sabe que alrededor del 40-60% de los genes exhiben empalmes alternativos.,
Gen Mecanismo de Splicing
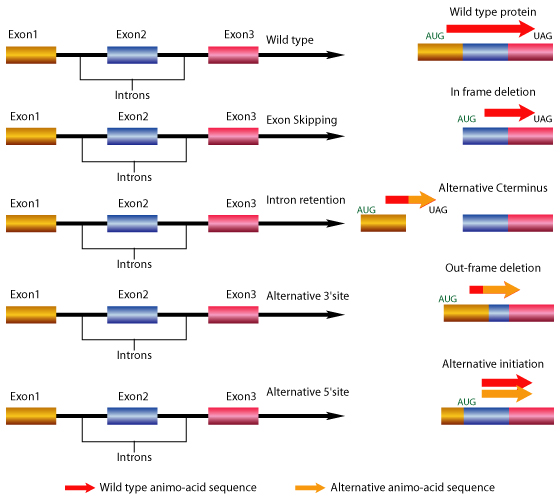
Hay varios tipos comunes de empalme de genes eventos. Estos son los eventos que pueden ocurrir simultáneamente en los genes después de que el ARNm se forme a partir del paso de transcripción del dogma central de la biología molecular.
omitir exón: este es el mecanismo de empalme genético más común conocido en el que se incluyen o excluyen exón(s) de la transcripción genética final que conduce a Variantes de ARNm extendidas o acortadas., Los exones son las regiones codificantes de un gen y son responsables de producir proteínas que se utilizan en varios tipos de células para una serie de funciones.
retención de intrones: un evento en el que se retiene a un intrón en la transcripción final. En humanos se ha notificado que el 2-5% de los genes retienen intrones. El mecanismo de empalme del gen retiene las porciones no codificantes (basura) del gen y conduce a una demornidad en la estructura y funcionalidad de la proteína.
sitio de empalme alternativo de 3′ y sitio de empalme de 5′: El Empalme de genes alternativo incluye la Unión de diferentes sitios de empalme de 5′ y 3′., En este tipo de empalme de genes, dos o más sitios de empalme alternativos de 5′ compiten por unirse a dos o más sitios de empalme alternativos de 3′.
Métodos de detección de variantes de empalme
El Empalme de genes conduce a la síntesis de proteínas alternativas que desempeñan un papel importante en la fisiología humana y la enfermedad. Actualmente, los métodos más eficientes para la detección a gran escala de variantes de empalme incluyen métodos de predicción computacional y análisis de microarray. La detección de variantes de empalme basada en Microarray es el método más popular actualmente en uso., La naturaleza altamente paralela y sensible de los microarrays los hace ideales para monitorear la expresión génica en un nivel específico de tejido, a nivel de todo el genoma. Los métodos basados en Microarray para detectar Variantes de empalme proporcionan una plataforma robusta y escalable para el descubrimiento de alto rendimiento del empalme genético alternativo. Un número de nuevas transcripciones de genes se detectaron utilizando métodos basados en microarray que no fueron detectados por ESTs utilizando métodos computacionales. Otro método comúnmente utilizado para descubrir nuevas isoformas de genes es RT-PCR seguido de secuenciación., Este es un enfoque poderoso y se puede usar de manera efectiva para analizar un pequeño número de genes. Sin embargo, solo proporciona una visión limitada de la estructura génica, es intensiva en mano de obra y no se escala fácilmente a miles de genes o cientos de tejidos.
desafíos en el diseño de Microarray para la detección de variantes de empalme
la detección de empalme genético basada en Microarray plantea algunos desafíos únicos en el diseño de sondas para isoformas que muestran un alto grado de homología., Para diferenciar entre estas isoformas, se utiliza un microarray que utiliza una combinación de sondas para exones y uniones exón-exón. Los eventos de omisión de exones u otras eliminaciones se pueden monitorear mediante el uso de sondas de unión. Por ejemplo, una sonda que abarca el exón 1 y el exón 3 del gen detectará el salto del exón 2 del gen que se traduce en una proteína.
Software para diseñar microarrays Variantes de empalme
AlleleID® diseña automáticamente sondas de unión, así como sondas intra-exón., Con AlleleID ® puede diseñar cualquier combinación de sondas para detectar cualquier tipo de Evento de empalme alternativo.