el cáncer se refiere a un grupo complejo y heterogéneo de enfermedades caracterizadas por la proliferación incontrolada y desordenada de células, que a menudo adquieren la capacidad de invadir otros tejidos. El cáncer suele originarse en células somáticas que, como consecuencia de una serie de mutaciones genéticas, evaden los mecanismos reguladores de la homeostasis tisular, como la inhibición del contacto entre células, las señales de diferenciación y la inducción de la muerte celular., Las mutaciones responsables de la transformación tumoral se refieren a dos grupos principales de genes, conocidos como proto-oncogenes (estimuladores del ciclo celular) y supresores tumorales (represores de la progresión del ciclo celular). Estas alteraciones funcionales pueden ocurrir como consecuencia de mutaciones de un solo nucleótido, pero también pueden ser causadas por modificaciones mayores en el material genético, como inserciones, deleciones, duplicaciones o translocaciones de un fragmento cromosómico. Estas anomalías en las células cancerosas se pueden utilizar como biomarcadores tumorales., La cuantificación de los cambios en el número de copias de genes o reordenamientos de genes es fundamental para nuestra comprensión de la biología tumoral, de ahí la importancia de las pruebas genéticas basadas en perfiles de citogenética molecular.
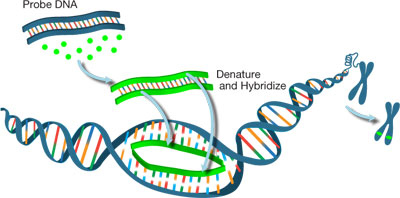
Figura 1: Sonda de etiquetado de ADN para análisis de peces.
¿Qué es un PEZ ?
la hibridación fluorescente in situ (FISH) es una técnica citogenética utilizada para la detección y localización de secuencias de nucleótidos (ADN o ARN) dentro de tejidos o células., Los peces se pueden utilizar para «mapear» el material genético en las células humanas, proporcionando información sobre la ubicación, la longitud y el número de copias de genes específicos o porciones cromosómicas. Requiere la síntesis de una sonda marcada fluorescentemente, que es una secuencia de ácido nucleico unida a una molécula reportera fluorescente, que luego se unirá (hibridará) a una secuencia objetivo específica. Las sondas FISH se pueden etiquetar por varios métodos (por ejemplo, traducción de nick, cebado aleatorio), a partir de diferentes entradas de ácidos nucleicos, como ADN/ARN genómico, cromosomas artificiales bacterianos (BACs) o cósmidos., el primer paso en el proceso FISH es inmovilizar las células cromosómicas de metafase o interfase que contienen el ADN objetivo en un portaobjetos de vidrio. A continuación, tanto la muestra de ADN como la sonda FISH son desnaturalizadas por calor. Cuando la sonda está en contacto con el material genético objetivo, se unirá específicamente a su secuencia complementaria en el cromosoma. Los híbridos formados entre las sondas y sus objetivos de secuencia se pueden visualizar utilizando un microscopio fluorescente (Figura 1)., En general, se pueden distinguir dos tipos de sondas FISH: las sondas de enumeración cromosómica (CEPs o CENs) que se dirigen a las regiones pericentroméricas de los cromosomas y se utilizan para enumerar cromosomas; e indicadores específicos de locus (LSI) que reconocen específicamente genes de interés.
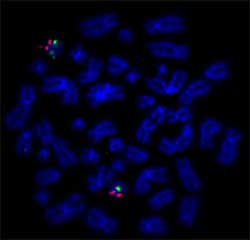
Figura 2: Ejemplo de resultados de FISH: Nick Translation DNA Labeling System 2.0 se utilizó para etiquetar la sonda de ADN BAC para TP53 con seebright® Orange 552 dUTP y la sonda de ADN BAC para centrómero 17 con seebright® Green 496 dUTP., Las sondas etiquetadas se hibridaron para propagarse en metafase. (Institut Universitaire du Cancer Toulouse Oncopole)
FISH tiene varias ventajas sobre otras técnicas citogenéticas clásicas, como el cariotipado con bandas G. Primero, tiene una resolución más alta (20-150KB vs 5Mb). Además, el FISH se puede aplicar a cromosomas metafásicos e interfásicos, lo que significa que las células no necesitan ser cultivadas durante varios días antes de que los cromosomas puedan prepararse para el análisis., Esto también implica que el FISH es adecuado para el análisis de diferentes tipos de muestras, incluidos los tumores sólidos y los tejidos incrustados en parafina fija con formalina (FFPE). Además, las sondas FISH pueden etiquetarse con diferentes fluoróforos, lo que permite el monitoreo simultáneo de múltiples sitios.
FISH-Ing aberraciones cromosómicas en cáncer
gracias a su versatilidad, FISH se puede utilizar para el análisis citogenético tanto de tumores sólidos (por ejemplo, cáncer de mama, cáncer de pulmón no microcítico, cáncer colorrectal) como hematológicos o cáncer de la sangre (por ejemplo, leucemia, linfomas, mieloma múltiple)., La detección de anomalías genéticas es útil no solo para el cáncer, sino también como una herramienta para analizar la predisposición genética y la información específica de la enfermedad, y para predecir un resultado quimioterapéutico.
FISH para el cáncer de pulmón
el cáncer de pulmón es la causa más comúnmente diagnosticada y la principal de muertes relacionadas con el cáncer. En particular, el cáncer de pulmón de células no pequeñas (NSCLC) representa ~80-85% de todos los cánceres de pulmón. Las mutaciones somáticas en los genes EGFR y ALK a menudo se asocian con el NSCLC., El EGFR (receptor del factor de crecimiento epidérmico) es una clase de receptores de tirosina quinasa cuya actividad está desregulada en diferentes tipos de neoplasias malignas epiteliales (incluido el cáncer de pulmón), ya que desempeñan un papel importante en la proliferación de células cancerosas, la angiogénesis y la metástasis. Por esta razón, las diferentes estrategias para interferir con la función del EGFR son comúnmente explotadas para la terapia de los pacientes. Los inhibidores de la actividad de la tirosina quinasa de estos receptores (por ejemplo, erlotinib, gefitinib) se utilizan ampliamente en los tratamientos clínicos de CPNM., Desafortunadamente, debido a la variedad de mutaciones genéticas subyacentes a la disfunción EGFR, algunos pacientes son insensibles a este tipo de tratamiento. De hecho, se pueden distinguir diferentes grupos de pacientes en función del tipo de alteración transportada por el EGFR, como la amplificación génica, la deleción o las sustituciones de un solo nucleótido, que pueden alterar su actividad de diferentes maneras (es decir, no a través del dominio tirosina quinasa). Como resultado, el número de copia EGFR determinado por FISH es uno de los biomarcadores utilizados para seleccionar la terapia correcta.,
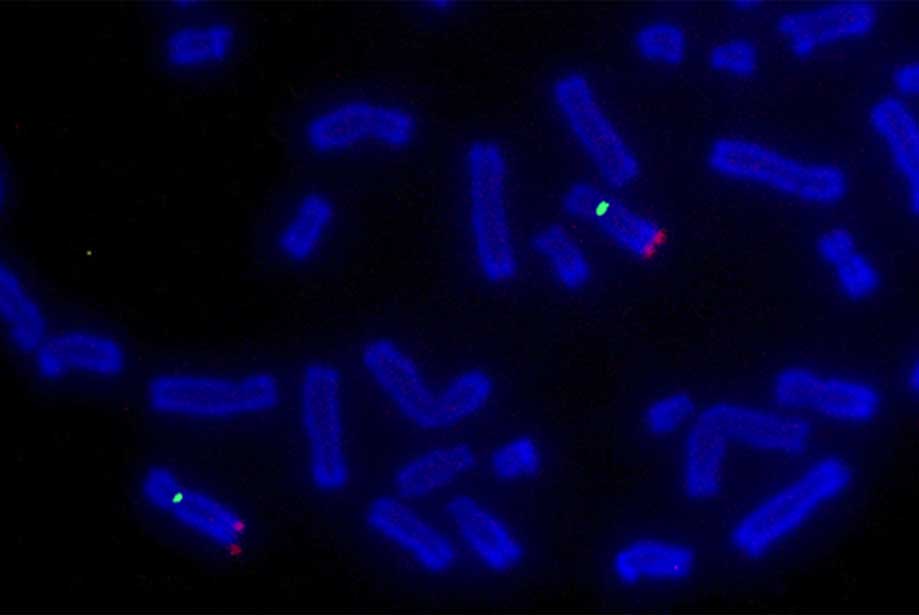
FISH se usa comúnmente para detectar inversiones o translocaciones en el gen ALK. El gen ALK se encuentra en el brazo corto del cromosoma 2 (2p23) y codifica para el receptor transmembrana de tirosina quinasa. ALK no debe expresarse en el pulmón adulto. Sin embargo, bajo condiciones patológicas, el gen ALK se rompe y fusiona su 3′ (que contiene el dominio tirosina quinasa) con el 5′ de otros genes. Este evento puede conducir a la activación incontrolada de las vías de señalización aguas abajo de ALK. La fusión más común ocurre con EML4, debido a una inversión en el brazo corto del cromosoma 2.,FISH es el método aprobado por la FDA para detectar inversiones o translocaciones en el gen ALK. Por lo general, las sondas dirigidas a las regiones 3′ y 5′ del gen están marcadas con diferentes fluoróforos: en los núcleos negativos, los colores aparecerán cerca unos de otros (a menudo superpuestos), mientras que en las células cancerosas las señales se dividirán como resultado del reordenamiento cromosómico (Figura 4).

Figura 3: Vista representativa de la translocación del gen ALK detectada por FISH., Las regiones 5′ y 3 ‘ del gen se visualizan con una fluorescencia verde y una roja respectivamente. A. núcleo de tipo salvaje. B. núcleo de células cancerosas, mostrando las sondas características divididas. C. núcleo de células cancerosas, mostrando las sondas características divididas. Una tercera sonda puede ser usada para definir el reordenamiento cromosómico que realmente ocurre.
Una tercera sonda dirigida al posible compañero de ALK podría confirmar y definir el reordenamiento cromosómico que ocurre en un paciente (figura 4C)., Los cánceres de pulmón que albergan mutaciones que conducen a la hiperactivación de ALK se pueden tratar con inhibidores de ALK como Crizotinib.
FISH para el cáncer de mama
esta es la neoplasia maligna más común en las mujeres y la segunda causa principal de muerte relacionada con el cáncer en todo el mundo. El cáncer de mama a menudo se caracteriza por anomalías en el estado del receptor, lo que lleva a una regulación ascendente de las vías de transducción celular responsables de la proliferación y supervivencia celular. En particular, se sabe que aproximadamente el 20-30% de los tumores de cáncer de mama sobreexpresan HER2/Neu, un miembro de la familia EGFR., Un tratamiento común en estos casos es Trastuzumab, un anticuerpo monoclonal humanizado aprobado por la FDA en 1998 para el tratamiento del cáncer de mama. Su mecanismo molecular exacto permanece para ser elucidado, pero este anticuerpo previene probablemente la activación de HER2 atando a su dominio extracelular. Además, parece inducir lisis de células tumorales estimulando la citotoxicidad celular dependiente de anticuerpos (CCDA). FISH, utilizando una sonda adecuada contra HER2, se puede utilizar para identificar copias adicionales del gen, un signo de que es más probable que responda al tratamiento con Trastuzumab.,
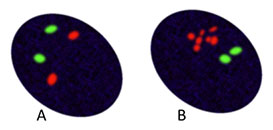
Figura 4: Vista representativa de células potenciales de carcinoma de mama. La señal HER2 se representa en rojo; La sonda centrómero 17 (Verde) se puede usar para enumerar el número cromosómico. A. HER2-carcinoma de mama no amplificado: dos centrómeros 17 y dos copias del gen HER2 como se esperaba. B. HER2-carcinoma de mama amplificado detección múltiple de HER2.
FISH para el cáncer de vejiga
es el quinto tumor maligno humano más común y el segundo tumor genitourinario más frecuentemente diagnosticado después del cáncer de próstata., Es una enfermedad poligénica, lo que significa que se ha asociado a múltiples anomalías genéticas, como mutaciones en los genes FGFR3, RB1, HRAS, TP53 o TSC1. Sin embargo, el evento de iniciación es probablemente una mutación en la región 9p21, que contiene el gen P16/CDKN2A. Además, las células cancerosas de vejiga se caracterizan por un grado elevado de inestabilidad cromosómica (NIC). La duplicación o segregación cromosómica disfuncional durante la mitosis causa reordenamientos y translocaciones del ADN, ganancia o pérdida de cromosomas completos (aneuploidía) o fragmentos cromosómicos., El desequilibrio resultante del material genético empeora después de cada ciclo celular. Los patrones genómicos consiguientes pueden estar relacionados con los diferentes estadios de desarrollo tumoral, siendo las formas más invasivas las que presentan mayor número de alteraciones citogenéticas.las alteraciones cromosómicas numéricas y estructurales que se encuentran en las células cancerosas de vejiga se pueden utilizar como marcadores tumorales.
específicamente, la detección simultánea de la variación del número de copias de los cromosomas 3, 7 y 17 y la eliminación de la región 9p21 (que contiene p16) por FISH utilizando cuatro sondas distintas es una práctica común., Este método es útil para proporcionar información sobre la progresión y la recidiva del cáncer.
FISH para leucemia linfocítica crónica (LLC)
el análisis de neoplasias hematológicas es uno de los ejemplos típicos de las ventajas de FISH para el análisis de muestras caracterizadas por un cariotipo variable y una baja actividad mitótica. LA LLC es la leucemia más común en adultos. No se ha asociado con una alteración genética recurrente específica., En cambio, de manera similar al cáncer de vejiga, se ha relacionado un panel de diferentes mutaciones con diferentes grados de gravedad de la enfermedad y se utilizan como indicadores predictivos del curso clínico del paciente. Los paneles FISH, en este caso, a menudo incluyen sondas para detectar la trisomía 12 y las deleciones 11q, 13Q y 17P., La deleción 11q, que en la mayoría de los casos se refiere al gen ATM, se encuentra en pacientes que muestran una progresión rápida del cáncer; la trisomía 12 se asocia con estadios avanzados de la enfermedad, resistencia a la quimioterapia y tiempos de supervivencia más cortos; la deleción 13q es la más frecuente y en general se asocia con un pronóstico más favorable; la deleción 17P a menudo incluye una deleción del gen TP53 y corresponde a estadio avanzado del tumor, con una tasa de supervivencia pobre.
Enzo Life Sciences es un líder mundial en tecnologías de etiquetado de ADN y ARN., Ofrecemos una gama de productos para sus necesidades de investigación genómica y cáncer. Para obtener un método simple y eficiente para generar ADN etiquetado, consulte nuestro kit de etiquetado de ADN Nick translation, así como una lista de nuestros dye-dUTPs fluorescentes SEEBRIGHT® y nuestro allylamine-dUTP. ¿No está seguro de si debe usar biotina o digoxigenina para el etiquetado de sondas ISH? Podemos ayudar! No olvide revisar el visor de espectros de Enzo para ver los perfiles de longitud de onda de excitación y emisión de tintes comunes y nuestros otros ensayos basados en células de fluorescencia., Mientras lo hace, consulte nuestra Nota técnica sobre los pros y los contras de FISH, aCGH y NGS y cómo usar allylamine-dUTP para el etiquetado de sondas de ADN de pescado. Para todas las preguntas e inquietudes relacionadas con cualquiera de nuestros productos, nuestro equipo de soporte técnico está aquí para ayudarlo.