¿cuántos genes en un genoma?
modo de lectura
ya hemos examinado la gran diversidad de tamaños de genoma en todo el mundo viviente (ver tabla en la viñeta sobre «¿qué tan grandes son los genomas?”). Como primer paso para refinar nuestra comprensión del contenido de información de estos genomas, necesitamos una idea del número de genes que albergan. Cuando nos referimos a genes estaremos pensando en genes codificadores de proteínas excluyendo la colección cada vez mayor de regiones codificantes de ARN en los genomas.,
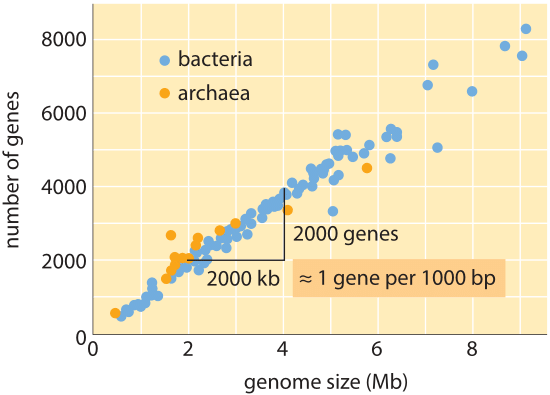
Figura 1: Número de genes como una función del tamaño del genoma. La figura muestra datos para una variedad de bacterias y arqueas, con la pendiente de la línea de datos confirmando la simple regla empírica que relaciona el tamaño del genoma y el número de genes. (Adaptado de M. Lynch, the Origins of Genome Architecture.,)
en todo el árbol de la vida, aunque los tamaños del genoma difieren hasta en 8 órdenes de magnitud (de <2 kb para el virus de la Hepatitis D (BNID 105570) a >100 Gbp para el Pez Luna jaspeado (BNID 100597) y ciertas flores de Fritillaria (BNID 102726)), el rango en el número de genes varía en menos de 5 órdenes de magnitud (desde virus como ms2 y bacteriófagos QB que tienen solo 4 genes hasta cerca de cien mil en el trigo). Muchas bacterias tienen varios miles de genes., Este contenido de genes es proporcional al tamaño del genoma y el tamaño de la proteína como se muestra a continuación. Curiosamente, los genomas eucariotas, que a menudo son mil veces o más grandes que los de los procariotas, contienen solo un orden de magnitud más genes que sus contrapartes procariotas. La incapacidad de estimar con éxito el número de genes en eucariotas basado en el conocimiento del contenido genético de procariotas fue uno de los giros inesperados de la biología moderna.,
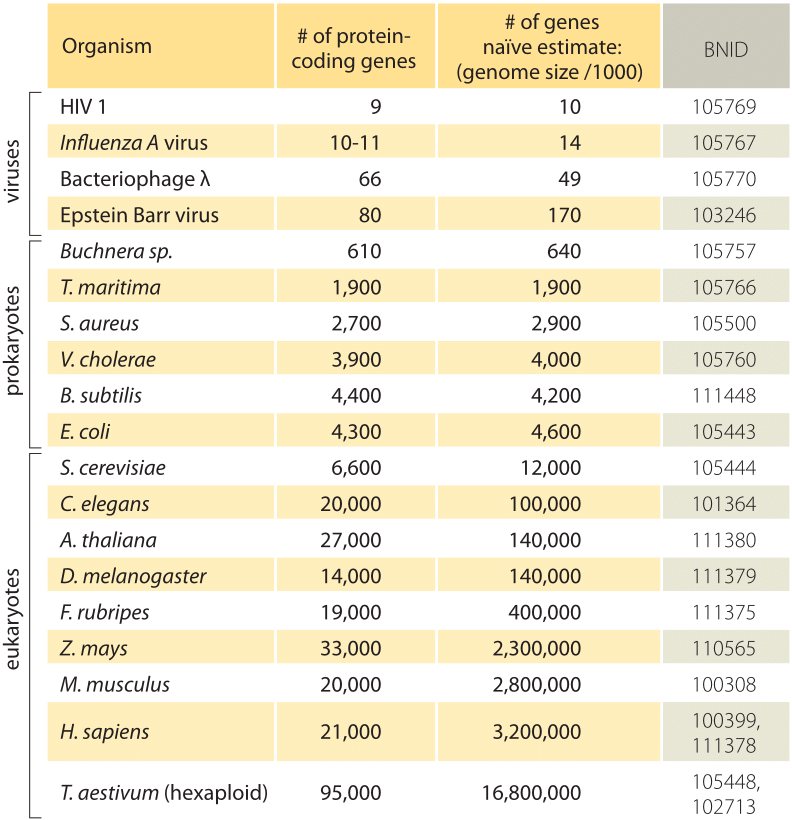
Tabla 1: una comparación entre el número de genes en un organismo y una estimación naïve basada en el tamaño del genoma dividido por un factor constante de 1000bp/gen, es decir, el número predicho de genes = tamaño del genoma / 1000. Uno encuentra que esta cruda regla de oro funciona sorprendentemente bien para muchas bacterias y arqueas, pero falla miserablemente para los organismos multicelulares.
la estimación más simple del número de genes en un genoma se despliega asumiendo que la totalidad del genoma codifica los genes de interés., Para avanzar más con la estimación, necesitamos tener una medida del número de aminoácidos en una proteína típica que tomaremos como aproximadamente 300, sin embargo, conscientes del hecho de que, al igual que los genomas, las proteínas vienen en una amplia variedad de tamaños, como se revela en la viñeta sobre ese tema, «¿cuáles son los tamaños de las proteínas?”. Sobre la base de esta escasa suposición, vemos que el número de bases necesarias para codificar nuestra proteína típica es aproximadamente 1000 (3 pares de bases por aminoácido)., Por lo tanto, dentro de esta mentalidad, el número de genes contenidos en un genoma se estima que es el tamaño del genoma/1000. Para los genomas bacterianos, esta estrategia funciona sorprendentemente bien como se puede ver en la tabla 1 y la Figura 1. Por ejemplo, cuando se aplica a la E. coli K-12, genoma de 4.6 x 106 bp, esta regla de oro conduce a una estimación de 4600 genes, que se puede comparar con el mejor conocimiento actual de esta cantidad que es 4225. Al pasar por una docena de bacterias representativas y genomas arqueales en la tabla, se observa un poder predictivo similar al 10%., Por otro lado, esta estrategia falla espectacularmente cuando la aplicamos a genomas eucariotas, resultando por ejemplo en la estimación de que el número de genes en el genoma humano debería ser de 3.000.000, una sobreestimación bruta. La falta de fiabilidad de esta estimación ayuda a explicar la existencia del grupo de apuestas Genesweep que tan recientemente como principios de la década de 2000 tenía personas apostando por el número de genes en el genoma humano, con las estimaciones de las personas que varían en más de un factor de diez.,
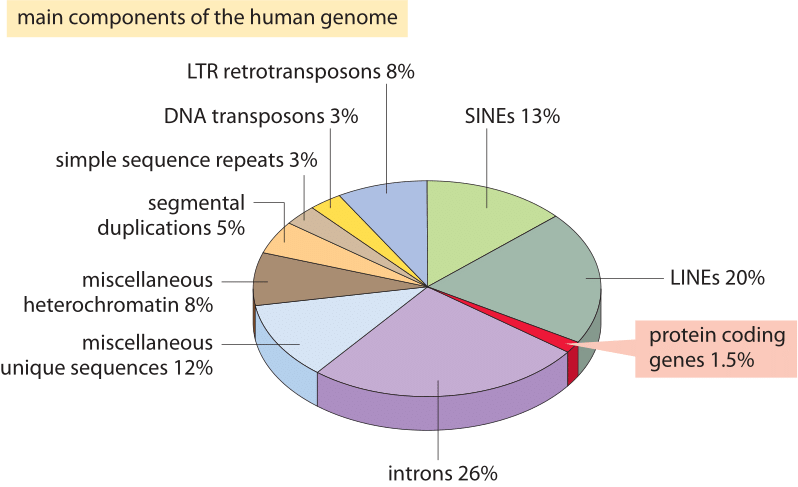
Figura 2: Los diferentes componentes de secuencia que componen el genoma humano. Alrededor del 1,5% del genoma consiste en el ≈20.000 secuencias codificantes de proteínas que son intercaladas por los intrones no codificantes, que constituyen alrededor del 26%. Los elementos transponibles son la fracción más grande (40-50%) incluyendo, por ejemplo, elementos nucleares intercalados largos (líneas), y elementos nucleares intercalados cortos (senos). La mayoría de los elementos transponibles son restos genómicos, que actualmente están extintos. (BNID 110283, adaptado de T. R. Gregory Nat Rev Genet., 9: 699-708, 2005 based on international Human Genome Sequencing Consortium. Secuenciación inicial y análisis del genoma humano. Nature 409: 860 2001.)
¿qué explica este espectacular fracaso de la estimación más ingenua y qué nos enseña sobre la información organizada en genomas? Los genomas eucariotas, especialmente aquellos asociados con organismos multicelulares, se caracterizan por una serie de características intrigantes que interrumpen la imagen de codificación simple explotada en la estimación ingenua., Estas diferencias en el uso del genoma se representan pictóricamente en la Figura 2 que muestra el porcentaje del genoma utilizado para otros fines que la codificación de proteínas. Como se evidencia en la Figura 1, los procariotas pueden compactar eficientemente sus secuencias codificantes de proteínas de tal manera que son casi continuas y dan como resultado que menos del 10% de sus genomas sean asignados a ADN no codificante (12% en E. coli, BNID 105750), mientras que en humanos más del 98% (BNID 103748) no es codificante de proteínas.,
el descubrimiento de estos otros usos del genoma constituyen algunos de los conocimientos más importantes sobre el ADN, y la biología en general, de los últimos 60 años. Uno de estos usos alternativos para los bienes raíces genómicos es el genoma Regulador, es decir, la forma en que grandes trozos del genoma se utilizan como objetivos para la Unión de proteínas reguladoras que dan lugar al control combinatorio tan típico de los genomas en los organismos multicelulares., Otra de las características clave de los genomas eucariotas es la organización de sus genes en intrones y exones, con los exones expresados siendo mucho más pequeños que los intrones intermedios y empalmados. Más allá de estas características, hay retrovirus endógenos, reliquias fósiles de infecciones virales anteriores y, sorprendentemente, más del 50% del genoma está ocupado por la existencia de elementos repetitivos y transposones, varias formas de los cuales quizás puedan interpretarse como genes egoístas que tienen mecanismos para proliferar en un genoma huésped., Algunos de estos elementos repetitivos y transposones todavía están activos hoy en día, mientras que otros han permanecido como una reliquia después de perder la capacidad de proliferar aún más en el genoma.
en conclusión, los genomas se pueden dividir en dos clases principales: compacto y expansivo. Los primeros son densos en genes, con solo alrededor del 10% de región no codificante y estricta proporcionalidad entre el tamaño del genoma y el número del genoma. Este grupo se extiende a genomas de hasta aproximadamente 10 Mbp, cubriendo virus, bacterias, arqueas y algunos eucariotas unicelulares., Esta última clase no muestra una correlación clara entre el tamaño del genoma y el número de genes, se compone principalmente de elementos no codificantes y cubre todos los organismos multicelulares.




